模拟计算测试——复达客户检测案例
2022年1月2日,本人接到西北某大学同学的计算需求,接到反馈后,第一时间与客户进行详细沟通,情况如下:
客户背景
客户是西北某大学的一位学生,其想得没食子酸、儿茶素、金丝桃苷、槲皮苷、积雪草酸、齐墩果酸、熊果酸、金丝桃苷、科罗索酸、α-淀粉酶、α-葡萄糖苷酶、胰脂肪酶、胆固醇脂酶之间的相互作用图,包括小分子与受体蛋白的结合位点,以什么方式结合在蛋白质的哪一个氨基酸上。能够得到活性成分与相应酶的结合位点的氨基酸组成,以及结合的方式。
样品名称
没食子酸、儿茶素、金丝桃苷、槲皮苷、积雪草酸分别和α-淀粉酶、α-葡萄糖苷酶
客户需求
其研究目的如下:
应用分子对接技术,推测单体成分没食子酸、儿茶素、金丝桃苷、槲皮苷、积雪草酸分别和α-淀粉酶、α-葡萄糖苷酶之间可能的结合位点。
推测单体成分齐墩果酸、熊果酸、金丝桃苷、科罗索酸和胰脂肪酶之间可能的结合位点。
推测单体成分齐墩果酸、熊果酸、科罗索酸和胆固醇脂酶之间可能的结合位点。
解决方案
1.受体结构准备
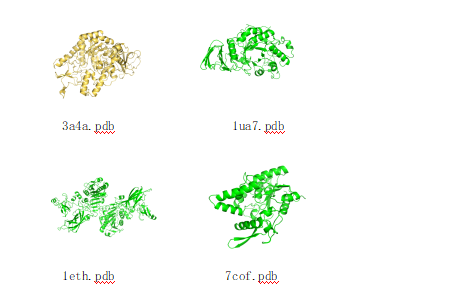
从PDB数据库中下载3a4a.pdb、1ua7.pdb、1eth.pdb、7cof.pdb结构文件。酿酒酵母α-葡萄糖苷酶PDB:3a4a.pdb,芽孢杆菌α-淀粉酶PDB:1ua7.pdb,猪胰腺-胰脂肪酶PDB:1eth.pdb,假单胞菌胆固醇脂酶PDB:7cof.pdb。用pymol对4个结构文件进行预处理得到初始结构,初始结构用AutoDock Tools 1.5.6 进行处理,计算Gaseigter电荷,并生成pdbqt文件用于分子对接。蛋白结构如下图:
2.配体结构准备
利用ChemBioDraw生成分子的2D文件,用ChemBio3D生成分子的PDB文件。在ChemBio3D中用MM2方法对小分子进行能量最小化,用于后续的分子对接,采用AutoDock Tools 1.5.6处理配体结构,生成pdbqt文件用于对接。
客户反馈
客户对数据结果满意,无论计算得到的数据还是我们提供的分析报告,以及后续的答疑都非常清晰严谨。






 792
792  792
792